Teams leaders: Jérôme Bonnet et Martin Cohen-Gonsaud
Synthetic Biology
Coordinator: Jérôme Bonnet
We work in the field of synthetic biology, the rational design and construction of new biological systems and functions. Synthetic biology builds on lessons from mature engineering disciplines and on exponential improvements in enabling technologies like DNA sequencing and synthesis.We are particularly interested in engineering genetic circuits allowing us to "program" cellular behavior, and to apply these tools to healthcare challenges, starting with diagnosis of disease.
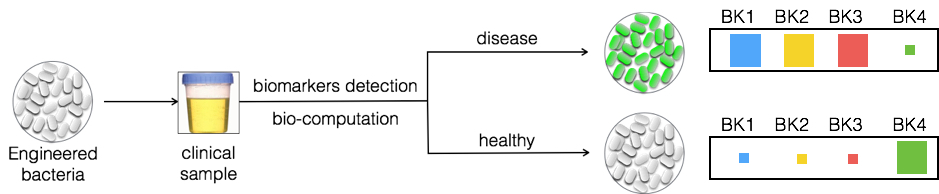
We aim at engineering bacterial biosensors, or "bactosensors" capable of detecting biomarkers of disease in clinical samples and to identfy pathological biomarkers signatures via biomolecular computation devices to perform early diagnosis of disease.


We are supported by the Inserm ATIP-Avenir Program, the Bettencourt-Schueller foundation and the ERC starting grant program.
M.tuberculosis structural biology
Coordinator: Martin Cohen-Gonsaud
The second field of research is centred on the structural biology of Mycobacterium tuberculosis proteins, the agent of Tuberculosis. We aim to use the structural biology data to understand the basics of M. tuberculosis pathogenesis, in particular to decipher the host-pathogen interactions.
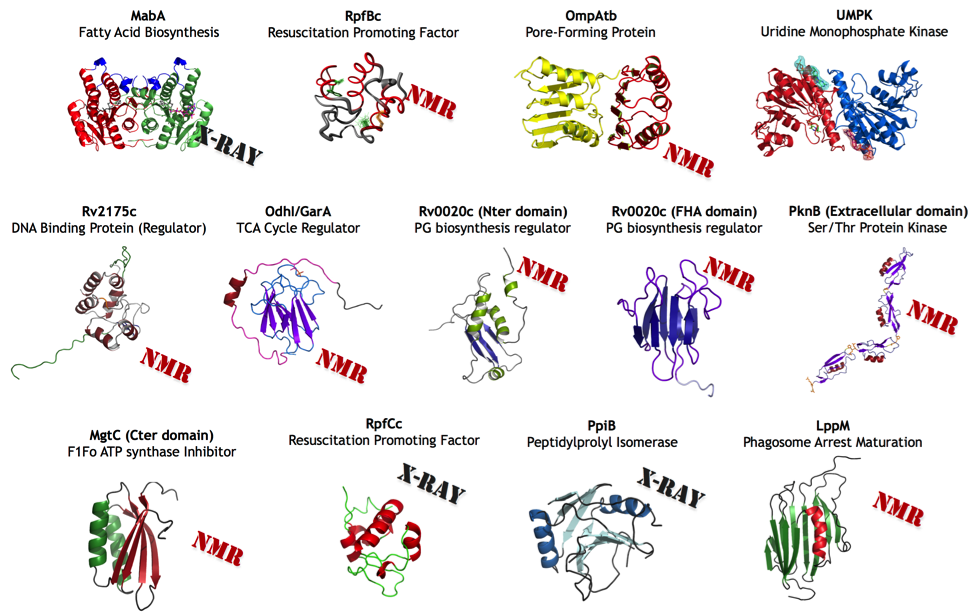
Mycobacterium tuberculosis protein structure gallery solved at the CBS