Nos projets visent à apporter une vision quantitative et intégrative des mécanismes moléculaires de régulation de la transcription chez les bactéries en combinant des approches in vivo et in vitro.
Terminaison de la transcription procaryote
N. Declerck, E. Margeat, C. Clerté, R. Vishwakarma
Ce travail se concentre sur les deux mécanismes utilisés par les bactéries pour terminer la transcription d'un gène (terminaison intrinsèque et Rho dépendante)
Nous utilisons une combinaison de techniques de molécule unique (smFRET, SPTPALM et spectroscopies de corrélation), nano-manipulation et biologie structurale pour étudier la dynamique des événements conduisant à la terminaison de la transcription chez les bactéries. Plus précisément, nos recherches portent sur:
1. Les antiterminateurs LicT et SacY de Bacillus subtilis, des protéines qui empêchent l'arrêt prématuré de la transcription par liaison à une boucle d'ARN qui chevauche un terminateur intrinsèque.
2. Le terminateur de transcription Rho, un hexamère ATP-dépendant doté d'activités hélicase et translocase, impliqué dans la dissociation des complexes d'élongation de transcription.
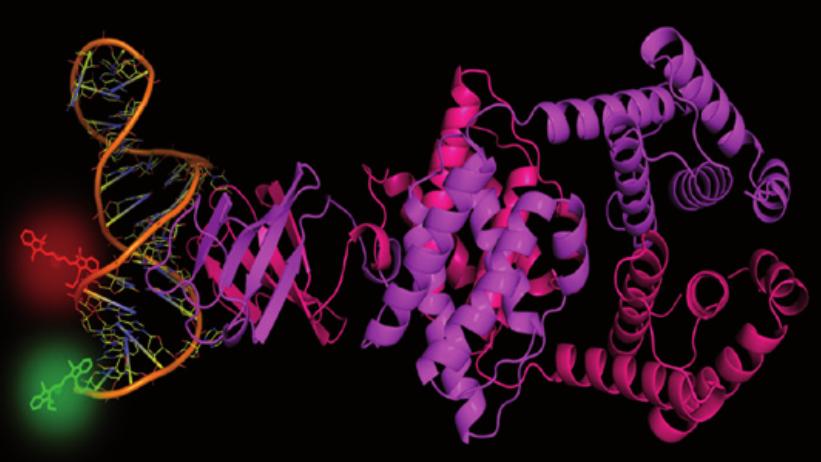
Cartoon representation of the LicT antitermination protein dimer, bound to its target RNA hairpin that has been labeled with a pair of fluorophores to monitor its conformational dynamics in a single pair FRET experiment.
Collaborations: H. Demene, F. Pedaci, M. Nollmann (CBS), M. Boudvillain (CBM, Orleans), N. Figueroa-Bossi (I2BC, Gif/Yvette), K. Brodolin (IRIM, Montpellier)
Adaptation des bactéries aux changements environnementaux
N. Declerck, A. Bourges, C. Clerté
Notre objectif est de comprendre les mécanismes moléculaires qui permettent l'adaptation des bactéries aux changements rapides de leur environnement. Nous cherchons notamment à confronter les résultats in vitro au comportement des protéines observées dans les cellules vivantes et les populations bactériennes.
Nous nous intéressons plus particulièrement aux répresseurs CggR et CcpN impliqués dans le contrôle de la glycolyse/néoglucogenèse chez B. subtilis. Des méthodes quantitatives très sensibles basées sur la mesure des fluctuations de fluorescence par microscopie bi-photonique nous permettent de déterminer la concentration intracellulaire, l'état d'oligomérisation et la diffusion de ces protéines régulatrices dans les cellules bactériennes vivantes. Combinées à des approches in vitro et in silico, nos travaux permettent d'établir un modèle de la réponse adaptative lors d'un changement de régime carboné. Une approche similaire est appliquée pour caractériser l'endonucléase Mrr responsable de l'activation de la réponse SOS dans les cellules E. coli exposées à de hautes pressions.

Live cell imaging of a B. subtilis strain expressing a PccgR-GFP transcriptional fusion under repression on malate.
Collaborations: S. Aymerich (INRA, Jouy-en-Josas), C. Royer (RPI, NY), O. Radulescu (UM, Montpellier)
G. Bellot, E. Margeat, N. Aissaoui, A. Mills
Nous utilisons les propriétés d'auto-assemblage des molécules d'ADN pour construire des architectures 3D à une résolution nanométrique. Notre objectif est de développer de nouveaux systèmes intelligents bio-inspirés pour aider à résoudre des problèmes d'intérêt fondamental et médical.
Nanotechnologie à base d'ADN. L'origami ADN est la méthode de référence pour concevoir des formes arbitraires et dynamiques à partir de fragments d'ADN. Notre objectif principal est d'explorer de nouveaux principes d'auto-assemblage pour la fabrication de matériaux à base d'ADN de plus en plus sophistiqués.
Protéines membranaires. La compréhension des mécanismes moléculaires à la surface de la membrane cellulaire nécessite de nouveaux outils expérimentaux à l'échelle du nanométre. Nous développons des nanostructures d'ADN pour faciliter les études structurales des protéines membranaires par cryo-EM et par microscopie de fluorescence sur molécule unique.
Nanomécanique bio-inspirée. Inspiré par les caractéristiques biomécaniques des assemblages protéiques, notre objectif est de construire des structures ADN de taille nanométrique qui présentent des fonctions et mouvements contrôlables. Ces nano-dispositifs artificiels sont utilisés comme capteurs pour detecter des stimuli spécifiques avec des applications dans des domaines tels que la biodétection et la biophysique.
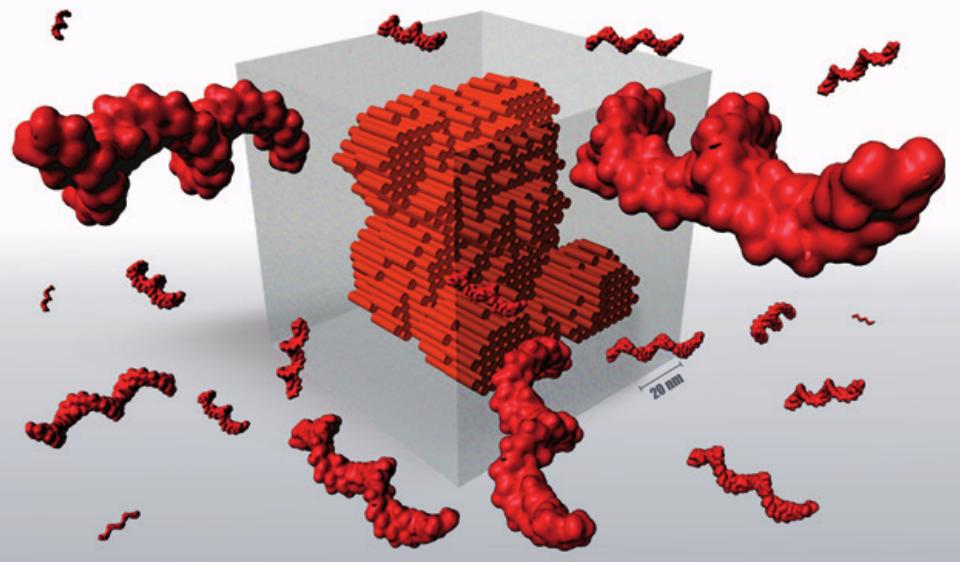
DNA self-assembly of 3D nanostructures from 1.5 million of nucleobases.
Collaborations: Y. Ke, (Georgia Tech, USA), S. Granier (IGF, Montpellier), S. Bidault, (ESPCI, Paris).

