G. Labesse, J-L. Pons, J. Gracy, V. Moreau, R. Salgado Ferreira, M. Schneider
La bioinformatique structurale vise à analyser et à prédire la structure des protéines à partir de séquences. Nous développons des outils afin d'améliorer la rapidité et la qualité des modèles théoriques que l'on peut construire. En outre, nous mettons en œuvre une interface intégrée reliant la modélisation de la structure des protéines et le criblage des ligands.
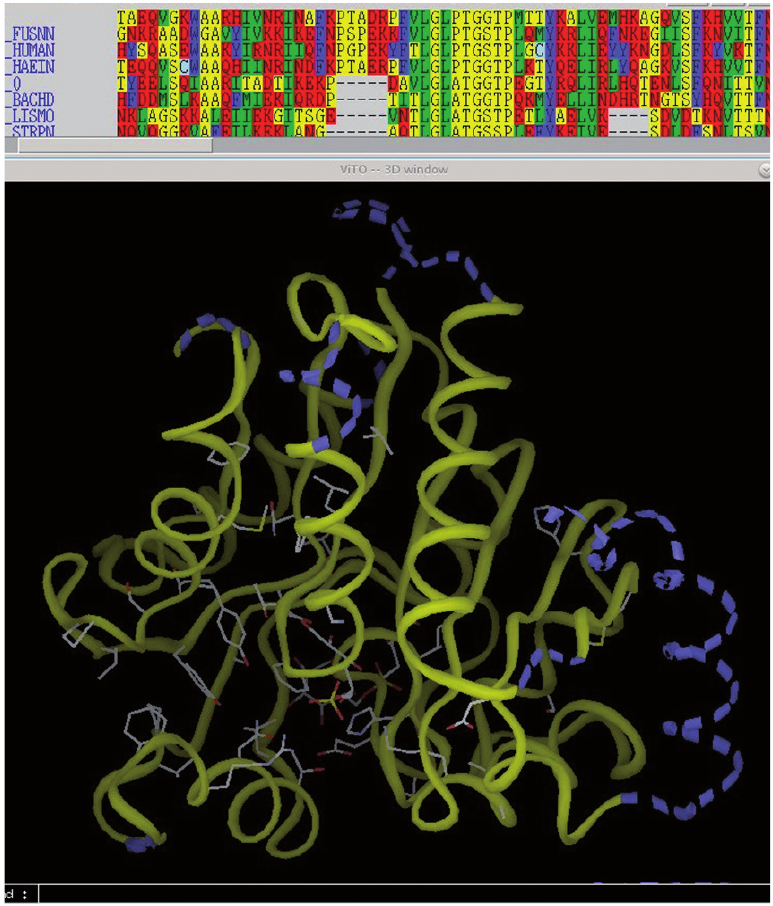
3D visualization and sequence editing using VITO for modeling distantly related proteins
La reconnaissance des repliements s'est avérée très efficace pour détecter des similitudes même faibles (15-25% d'identité de séquences). Néanmoins, l'alignement et la construction de modèles corrects à partir de structures faiblement homologues nécessitent encore une expertise et un travail importants. La mise en œuvre de nouvelles approches de raffinement est nécessaire. Parallèlement, l'amarrage comparatif a montré une robustesse inattendue même à un faible niveau de conservation de séquence, ce qui a permis certaines annotations fonctionnelles. Nous concentrons maintenant notre développement méthodologique sur l'amélioration de l'ancrage des ligands par l'exploration de conformations multiples.
Collaborations : P. Tuffery (Paris Diderot, Paris)
References : Pons & Labesse, NAR, 2009 ; Martin et al., NAR, 2006

