Thème 1 : Récepteurs nucléaires et corégulateurs
A. le Maire, P. Germain, W. Bourguet
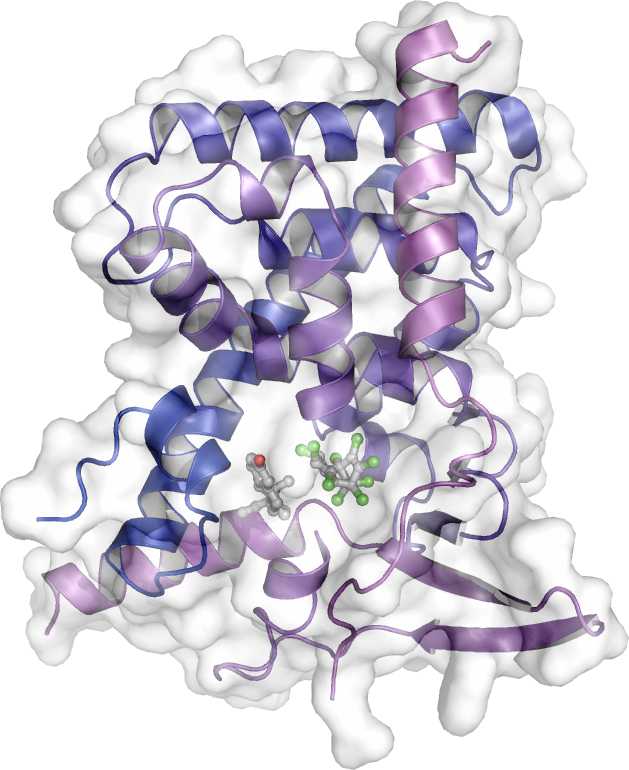
La régulation précise de l'expression génique nécessite l'activation et la répression de la transcription de l'ADN. Un certain nombre de maladies, y compris certains cancers, proviennent d'une répression dysfonctionnelle. L'objectif global de ce projet est d'obtenir des informations sur les mécanismes généraux régissant la (dé)régulation de la répression génique par les récepteurs nucléaires.
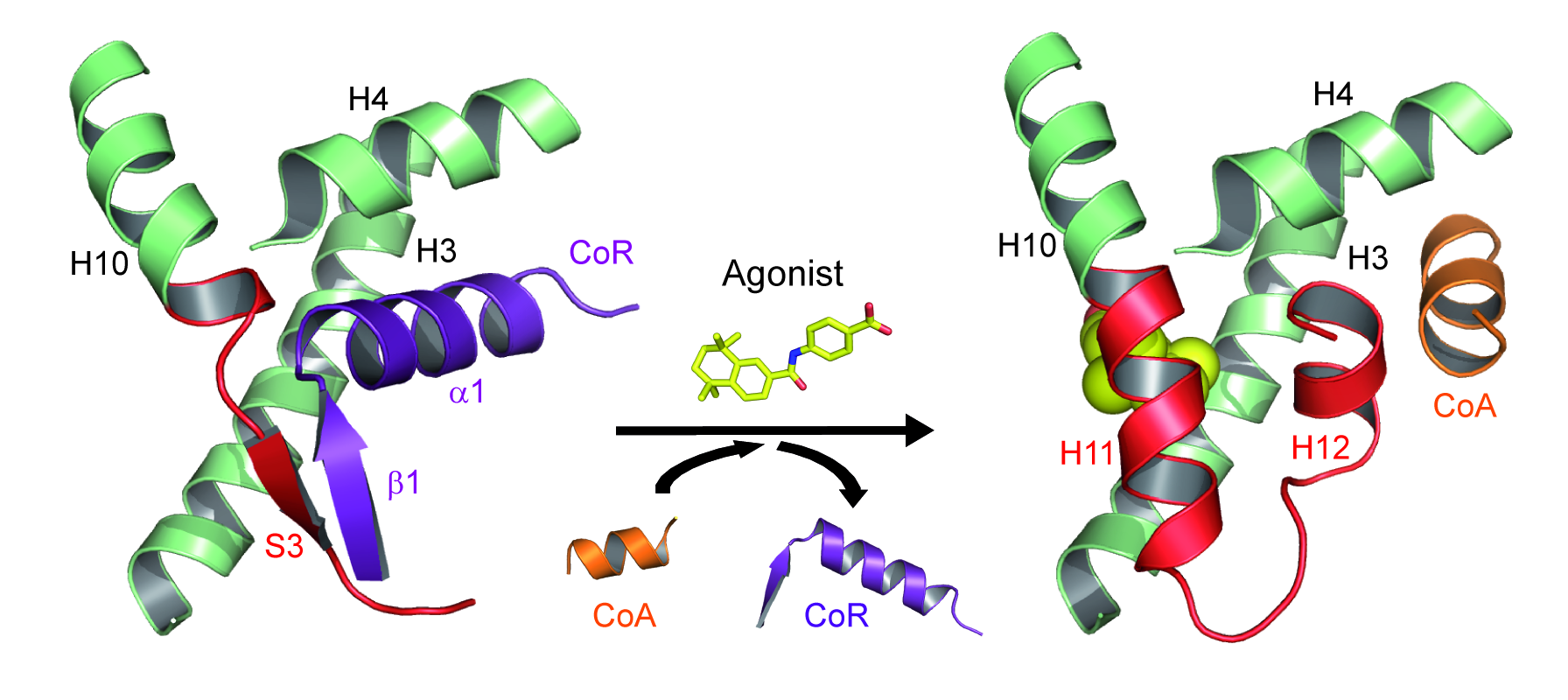 Association et dissociation des coregulateurs de RAR après liaison du ligand (CoA, coactivateur ; CoR, corépresseur).
Association et dissociation des coregulateurs de RAR après liaison du ligand (CoA, coactivateur ; CoR, corépresseur).
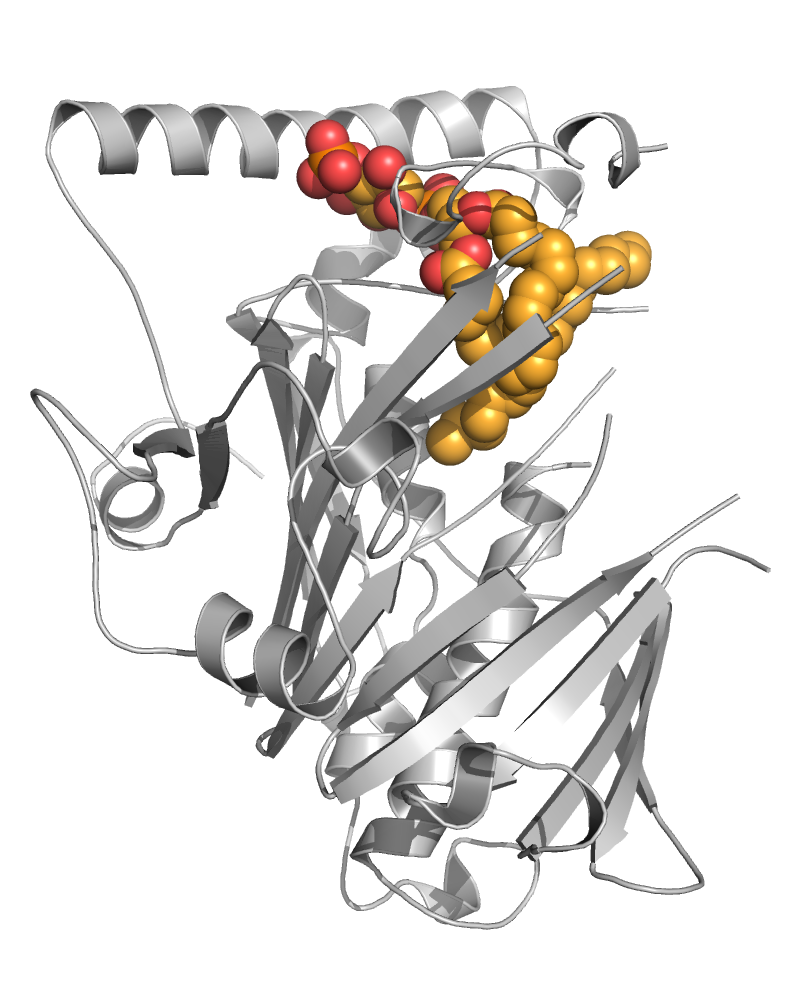
Certains récepteurs nucléaires, y compris ceux de l'acide rétinoïque (RAR) et de l'hormone thyroïdienne (TR) agissent comme des répresseurs transcriptionnels en recrutant des complexes corépresseurs sur leurs gènes cibles. Cependant, le rôle précis de l'inactivation des gènes reste encore mal compris par rapport à celui de leur activation, et plusieurs maladies (par exemple la leucémie promyélocytaire aiguë et les carcinomes rénaux) ont pour origine des interactions aberrantes entre des formes modifiées de récepteurs nucléaires et de corépresseurs. Les objectifs de ce projet sont (i) de révéler les mécanismes de la répression transcriptionnelle médiée par RAR et TR (sauvages et mutants), (ii) de déchiffrer comment cette activité de silencing peut être modulée par des ligands naturels et pharmacologiques, et (iii) fournir des outils moléculaires pour étudier l'importance de cette répression dans les processus physiologiques, développementaux et pathologiques. La force de notre projet réside dans la synergie qui émerge de la combinaison d'approches biophysiques, structurales, cellulaires, génomiques et in vivo.
Principaux collaborateurs : G. Benoit (ENS Lyon), F. Flamant (IGFL Lyon), N. Ghyselinck (IGBMC Illkirch), P. Veber (LBBE Lyon), M. Privalsky (UC Davis USA), AC. Figueira (CNPEM Brasil), A. Yunes (Boldrini Brasil).
Références : le Maire et al., Nat. Struct. Mol. Biol., 2010 ; Germain et al., Chem. Biol., 2009 ; Nadendla et al., Plos One, 2015 ; Sharman et al., NAR, 2011