Etude sur les boucles cis régulatrices de la chromatine
Les boucles cys régulatrices de la chromatine apparaissent avant les TADs et l'activation des gènes, et sont indépendantes du destin cellulaire durant le développement
Sergio Martin Espinola, Markus Götz, Jean-Bernard Fiche, Maelle Bellec, Christophe Houbron, Andrés M. Cardozo Gizzi, Mounia Lagha, Marcelo Nollmann
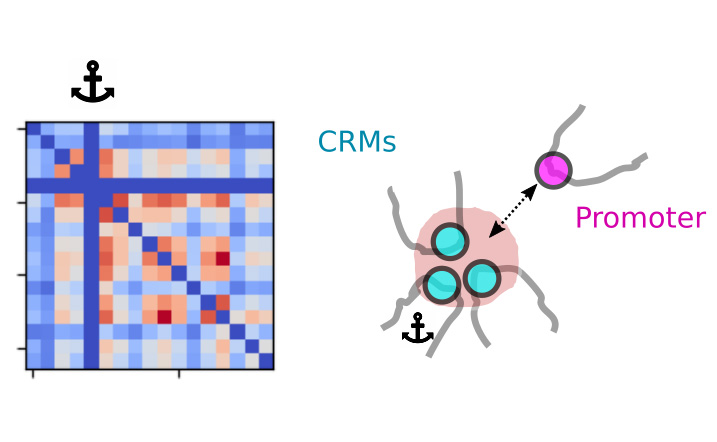
Au cours du développement, les cellules naïves acquièrent progressivement des destins cellulaires distincts, grâce à des mécanismes sophistiqués de régulation génique spatio-temporelle précise. On pense que l'acquisition du destin cellulaire repose sur l'interaction spécifique de modules de régulation *cis* distants (par exemple, des amplificateurs, des silencieux) (CRM) et de promoteurs cibles. Cependant, l'interaction précise entre la structure de la chromatine et l'expression des gènes n'est toujours pas claire, en particulier dans les cellules individuelles des organismes de développement multicellulaires. Dans ce cas, nous utilisons Hi-M, une approche de génomique spatiale des cellules uniques, pour détecter systématiquement les interactions entre les promoteurs CRM et les boucles dans les domaines d'association topologique (TAD) au cours du développement de la drosophile. En comparant les boucles régulatrices *cis* dans d'autres types de cellules, nous montrons que la proximité physique n'indique pas nécessairement les états de transcription. En outre, des analyses multidirectionnelles ont révélé l'existence d'interactions locales entre plusieurs CRM distants pour former des hubs. Nous avons constaté que les boucles et les centres de CRM sont établis au début du développement, avant l'émergence des TAD. De plus, les hubs de CRM sont formés par l'action du facteur de transcription pionnier Zelda et précèdent l'activation transcriptionnelle. Notre approche offre une nouvelle perspective sur le rôle des interactions entre le CRM et les promoteurs dans la définition des états d'activation et de répression transcriptionnelle, ainsi que des types de cellules distincts.
https://www.nature.com/articles/s41588-021-00816-z