Un modèle d'analyse hors-équilibre permet de déduire les vitesses relatives d'initiation et d'élongation de la traduction à partir de données de profilage ribosomique
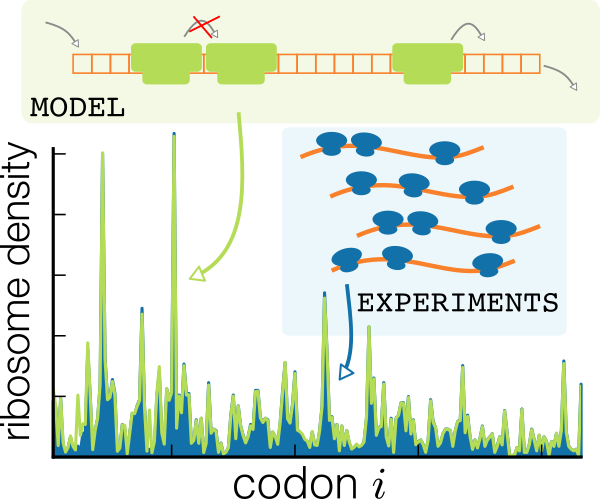
Un des plus grands challenges de la modélisation de la traduction des ARN messagers est d'identifier les caractéristiques des séquences codantes qui déterminent la vitesse de synthèse protéique. Des progrès techniques récents comme le profilage ribosomique permettent d'analyser la traduction à l'échelle du codon et de rendre plus accessibles les études quantitatives sur l'efficacité de transcription. Nous avons développé une méthode appelée NEAR qui intègre les données expérimentales de profilage ribosomique et un modèle hors équilibre bien connu de trafic ribosomique. Notre approche fournit des informations biologiques sur le contrôle du trafic et déduit la cinétique des ribosomes sur chaque transcrit. L'analyse des données de profilage des ribosomes chez la levure, montre que l'initiation et l'élongation de la traduction sont proches de leurs optima et que le trafic est minimisé au début du transcrit pour favoriser le recrutement des ribosomes. Nos travaux fournissent de nouvelles mesures de l'efficacité de l'initiation et de l'élongation de la traduction, soulignant l'importance de mesurer séparément ces deux étapes de traduction.